ポイント
- 光合成の電子伝達を担う鉄イオウタンパク質の全原子構造を世界で初めて、中性子結晶構造解析を用い高精度で決定
- タンパク質中のぺプチド結合の平面構造について新しいモデルを提唱
- 新しいモデルを用いたタンパク質の立体構造解析により、生命科学研究の進展に寄与
概要
国立研究開発法人量子科学技術研究開発機構(理事長 平野俊夫)量子生命・医学部門量子生命科学研究所構造生物学研究チームの玉田太郎チームリーダー、花園祐矢博士研究員(現、国立大学法人東京医科歯科大学難治疾患研究所先端分子医学研究部門 助教)、平野優主幹研究員、国立大学法人京都大学大学院理学研究科の三木邦夫名誉教授、竹田一旗准教授、国立大学法人茨城大学フロンティア応用原子科学研究センターの日下勝弘教授は共同で、光合成細菌1)の光合成電子伝達を担うタンパク質である高電位鉄イオウタンパク質(HiPIP)2)の高精度全原子結晶構造解析3)に成功しました。水素原子の観察に優れた中性子の特徴を生かして水素原子の位置を精密に決定し、タンパク質の立体構造形成に大きく影響するペプチド結合4)の平面構造について新たな構造モデルを提唱しました。
生命活動の中心的役割を担うタンパク質は、数十から数万のアミノ酸がペプチド結合により鎖状につながった分子です。一般にペプチド結合は平面構造が最も安定であり、タンパク質分子中のペプチド結合もすべて同じ平面構造であるとの仮定の下で、これまではタンパク質の構造が議論されてきましたが、その仮定を実際に確かめた研究例はありませんでした。なぜなら、タンパク質分子を構成する原子の約半分が水素原子のため、タンパク質の構造を決定する際に主に用いられるX線結晶構造解析という手法だけでは、高い精度で観測することができないためです。
そこで、研究グループは水素原子を直接観察できる中性子結晶構造解析という手法に着目しました。この解析を行うためには、極めて高品質かつ、通常X線結晶構造解析で用いられるサイズの約1万倍という大きな結晶試料を作成することが必要でしたが、これらの課題を克服し、水素原子を直接観測することができました。その結果、ペプチド結合はすべて同じ平面構造を取るという仮定に基づいた従来モデルを用いることなく、HiPIPの全原子構造を1.2Å(オングストローム。1Åは100億分の1メートル)分解能という極めて高い解像度で決定することに成功しました。その結果、ペプチド結合は周囲の環境によって多様な構造をしていることを世界で初めて明らかにし、ペプチド結合の新たなモデルを提唱しました。
今回明らかにしたペプチド結合の構造についての知見は、電子伝達機能などタンパク質の機能発現のメカニズムを理解する上で重要です。また、今回提唱する新しいモデルに基づいてタンパク質の立体構造を精密に捉えることで、生命科学研究の進展に貢献し、研究で得られた知見は、医学、薬学、工学分野に幅広く応用されることが期待できます。
本研究は、国立研究開発法人科学技術振興機構の光・量子融合連携研究開発プログラム(課題名:中性子と放射光の連携利用によるタンパク質反応プロセスの解明、三木邦夫)、戦略創造研究推進事業・さきがけ(課題番号:JPMJPR17G7、平野優)、文部科学省の光・量子飛躍フラッグシッププログラム(Q-LEAP)(課題番号:JPMXS0120330644、玉田太郎)、日本学術振興会科学研究費補助金 若手研究(B)(課題番号:JP15K18494、平野優)、研究活動スタート支援(課題番号:JP20K22642、花園祐矢、課題番号:JP25891030、平野優)および基盤研究(C)(課題番号:JP20K06523、三木邦夫)の助成を受けたものです。中性子回折実験は大強度陽子加速器施設(J-PARC)の物質・生命科学研究施設(MLF)の茨城県生命物質構造解析装置(iBIX)を用いて実施し、X線回折実験は高エネルギー加速器研究機構(KEK)のフォトンファクトリーにて実施しました。
本研究成果は、米科学誌Scienceの姉妹誌であるScience Advancesのオンライン版に2022年5月21日(土)3:00(日本時間)に掲載されました。
研究の背景
私たちの体の15~20%はタンパク質からできています。その種類は約10万種と言われており、微生物からヒトまで、すべての種において生命活動を担っている分子です。タンパク質は極めて小さくかつ精密な「分子機械」で、これがどのように生命反応を精妙に制御しているのかを理解するためには、その分子の立体構造を原子・電子レベルで正確に決定することが必要です。
タンパク質の立体構造は複雑で、20種類のアミノ酸がペプチド結合により鎖状につながり、折り畳まることで形作られます。ペプチド結合は平面性を有する場合が最も安定な構造をとることができ、その平面性を保ちながら短いもので数十、長いものでは数万のアミノ酸が折り畳まれていると考えられています。タンパク質の構造を最も精度よく決定できる方法は結晶構造解析という手法ですが、この手法においても、観測に必要な高い解像度を得るための実験データ数を十分に得ることができません。そのため、タンパク質中のすべてのペプチド結合は同じ構造を取っているという仮定に基づいて取り扱わざるを得ないのが現状です。また、構造決定に主に用いられているX線結晶構造解析という手法では、タンパク質分子を構成する原子の約半分を占める水素原子からの回折が非常に弱いため、タンパク質の安定性や構造形成に重要な水素原子を十分に高い精度で観測できません。そのため、ペプチド結合の構造の詳細を理解することはこれまで極めて困難でした。
研究の内容
そこで研究グループは、水素原子の観測に優れる「中性子結晶構造解析」に着目しました。しかしこの手法は、計測試料となるタンパク質の結晶の作製に非常に高度な技術を必要とします。中性子はX線に比べるとビームの強さが格段に弱いため、通常X線結晶構造解析で用いる結晶の約1万倍の大きさの結晶を作る必要があるためです。研究グループは、これまでに他の研究試料の中性子結晶構造解析にも複数成功しており、蓄積したノウハウ・知見を数多く有しています。その際に得た知見を活かしてHiPIPの高品質な大型結晶を作製しました。さらに、高分解能で観測するためには低温条件での測定が最適である反面、結晶試料の凍結条件によっては結晶の質を劣化させてしまうため、結晶試料を均一に凍結して劣化を防ぐ条件を検討し、低温条件下で結晶の質を損ねることなく高分解能での観測に成功しました。
中性子回折データ収集は、大強度陽子加速器施設(J-PARC)の物質・生命科学実験施設(MLF)内にある茨城県生命物質構造解析装置(iBIX)を用いて行いました。さらに、同一結晶から放射光施設(高エネルギー加速器研究機構のフォトンファクトリー)においてX線回折データを収集し、それを組み合わせた構造精密化によって、最終的に1.2 Å分解能という非常に高い解像度でHiPIPの立体構造を決定しました。有機化合物のようなタンパク質分子に比べて分子量が小さい分子の結晶構造解析は高い解像度で構造が決定されるため、その情報をモデルとして、タンパク質中の水素原子が関わる結合の長さや角度の情報を決定するのが一般的です。しかしながら小さい分子の構造情報が、タンパク質のような大きい分子の実験データを真に反映しているとは言えません。今回、解像度の高い中性子回折データが得られたことで、小さい分子の結晶構造から得られたモデルを用いることなく、HiPIPの結晶構造を精密に評価することができました。
タンパク質の構造の安定性や機能の発現には水素結合が深くかかわっています。水素結合は通常1つのドナー(電子を与える役目)とアクセプター(電子を受け取る役目)の組み合わせから構成されます。そのような水素結合においては、ドナー側のアミド基(N-H)とアクセプターの酸素原子間の角度(∠N-H...O)は小さい分子の構造から得られたモデルを使用した時より大きな値となるものが多く(図1A)、アミド基の水素原子(アミドプロトン)の原子核の位置はアクセプターの方へ向いていました(図1B)。よって、これまで同一と扱われてきたアミドプロトンは実際にはモデルからずれた箇所にも存在し、ペプチド平面(次項で詳しく解説します)からずれたアミドプロトンを多数観測しました。
一般にペプチド結合の平面構造は(図2A中の薄青い面)、アミドプロトンの構造情報がほとんど得られていないため、それ以外の炭素原子と窒素原子を用い議論されています(図2A中の黒点線囲み)。今回はアミドプロトンの位置を実験で正確に決定できたため、アミドプロトンを含む平面構造に着目しました(図2A中の赤い面)。その結果、アミドプロトンの位置は、水素結合のアクセプターである酸素原子の側にシフトしていることが明らかとなりました(図2B)。したがって、ペプチド結合の平面構造は、タンパク質内部で形成している水素結合により影響を受けて変化していることが明らかとなりました。このように、従来は画一的に平面構造を取っていると考えられていたペプチド結合が、タンパク質内の電子状態によってその平面性が影響を受けて多様な構造を取っていることを、本研究によって世界で初めて実験的に明らかにすることができました(図2C)。
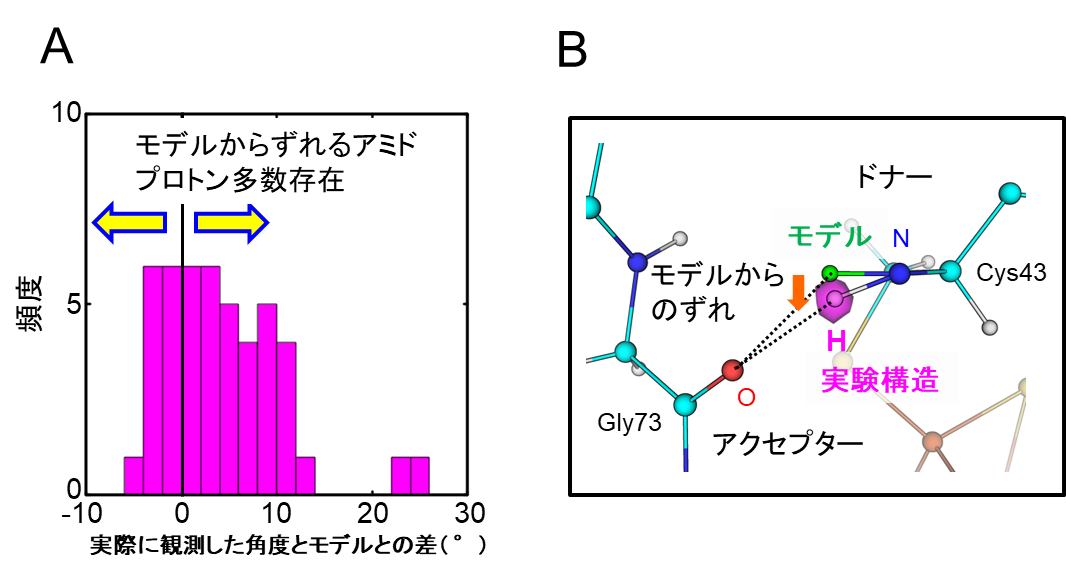
図1.モデルを使用せず実験データから直接決定したアミドプロトン
A:ドナー原子とアクセプター原子間の角度(∠N-H...O)の実験データとモデルとの差を示す。モデルからずれるアミドプロトンが多数存在し、モデルより大きい角度(右矢印)の頻度が高いことがわかる。B:実測した水素原子(実際は重水素原子)。マゼンタが原子核散乱長密度分布5)で水素(重水素)原子を除いて計算したもの。モデル(緑)に比べて角度は大きく、アクセプターの方を向いている。
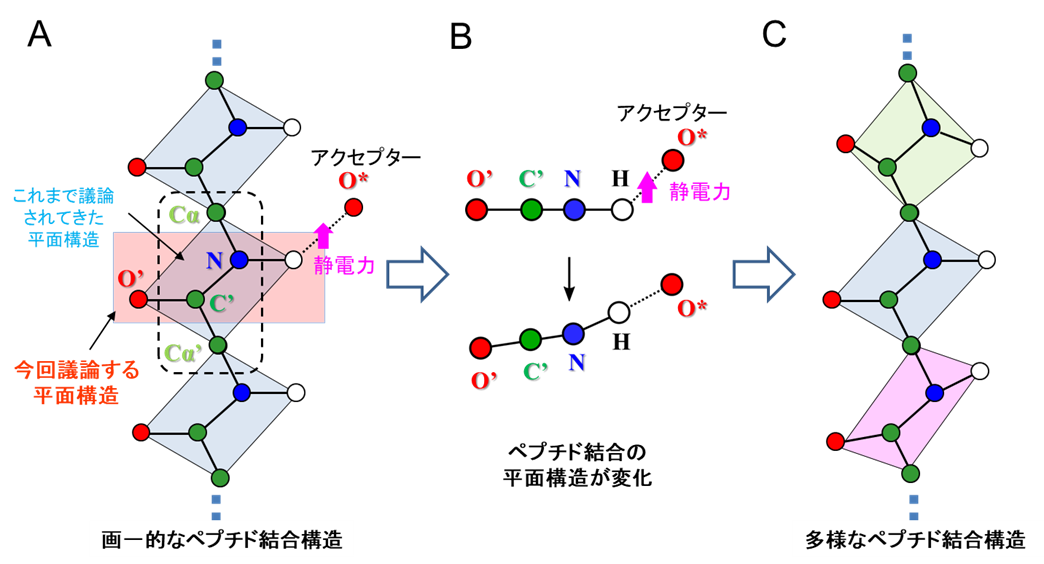
図2.ペプチド結合の平面性
A:タンパク質は青色で示されるペプチド平面が画一的な平面構造をとって折り畳まることで立体構造を形成するとこれまで考えられてきた。今回、実験的に確認したアミドプロトンは静電力6)によってアクセプター原子の方にシフトし、窒素原子が青色のペプチド平面から浮き上がり、赤色で示す別の平面性に影響していた。B:赤い平面を横から見た図。上はモデル、下は実験構造を示す。下の方が水平状態というよりは面が緩い角度で折れた状態を示している。C:実際にはペプチド結合は多様な構造を取っている。
今回明らかにしたペプチド結合の多様性は、タンパク質の機能にも関係していると考えられます。HiPIPは分子の中心に鉄イオウクラスター7)と呼ばれる金属補因子を含んでいます(図3A)。HiPIPは光合成細菌における光合成において、反応中心複合体に電子を渡しますが、その電子伝達を直接的に担うのが分子中に存在する鉄イオウクラスターです。
HiPIPは鉄イオウクラスターに電子が1個多い状態(還元型)から、反応中心に電子を1個渡すことで電子の1個少ない状態(酸化型)に変化し電子伝達反応が進行します。この酸化型と還元型の立体構造を見てみると、鉄イオウクラスターとそれを取り囲むアミノ酸との相互作用においてペプチド結合の構造に差が観測されました。鉄イオウクラスター近傍のアミノ酸(Cys75)において、酸化型では別のアミノ酸(Trp74)と相互作用していることが明らかになりました(図3B)。一方、還元型(京都大学のグループが2016年に発表した世界最高分解能のX線結晶構造解析の結果)では、鉄イオウクラスターを構成するイオウ原子の1つ(S3)と相互作用していることが示され(図3C)、電子の1個多い還元状態の鉄イオウクラスターを水素結合により安定化していると考えられます。酸化型と還元型のわずかな構造の違いで、HiPIPの電子伝達が制御されていると考えられます。
鉄イオウクラスターはヒトを含めた動物、植物などすべての生命体が持つものです。鉄イオウクラスターを利用した電子伝達反応の仕組みを理解することができれば、様々な生命現象の理解にもつながります。
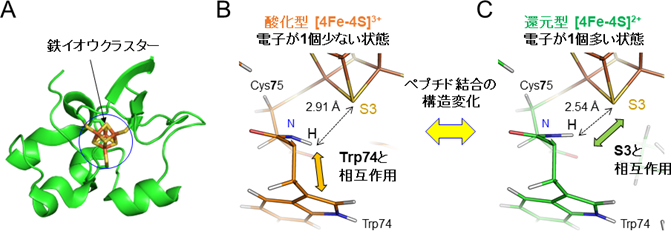
図3.HiPIPの鉄イオウクラスターを取り囲むアミノ酸の酸化型と還元型でのペプチド結合構造の違い
A: HiPIPは分子の中心に鉄イオウクラスターを含んでいる。B:酸化型の鉄イオウクラスターの近くに位置するアミノ酸(Cys75)は鉄イオウクラスター中のイオウ原子(S3)ではなく、別のアミノ酸(Trp74)と相互作用している。C:それに対し、還元型ではS3と相互作用している。
研究の成果と今後の期待
今回、1.2Å分解能という極めて高い解像度でHiPIPの中性子結晶構造解析に成功しました。世界で初めて、従来のモデルを用いることなく実験的に解析を実現したことで、従来モデルから外れた位置に存在する水素原子の位置を精密に決めることができました。本成果は、タンパク質中のペプチド結合は画一的な平面構造を取っているのではなく、タンパク質の電子状態によってペプチド結合の平面性が影響を受けて多様な構造を取っていることを世界で初めて明らかにし、新たなモデルを提唱するものです。また、ペプチド結合の多様性が鉄イオウクラスターの電子伝達反応を制御していることを示しており、精密な構造解析が機能のメカニズムを理解するうえで重要であることを示しました。
鉄イオウクラスターは光合成だけでなく、呼吸、酵素反応、遺伝子代謝など様々な生体反応に関わっていることが知られています。今回の成果を適応することで、HiPIPにとどまらず、上述の様々な生体反応の理解を深めることができます。さらに、今回明らかにしたペプチド結合の平面構造の新たなモデルを用いた構造解析により、数多くのタンパク質の精密な立体構造の決定が可能となります。その成果は医学、薬学、工学分野において幅広く応用されることが期待されます。
論文掲載情報
雑誌名:Science Advances
論文タイトル:Revisiting the concept of peptide bond planarity in an iron-sulfur protein by neutron structure analysis
著者:Yuya Hanazono, Yu Hirano, Kazuki Takeda, Katsuhiro Kusaka, Taro Tamada, Kunio Miki
DOI:10.1126/sciadv.abn2276
用語解説
1)光合成細菌
光合成を行う細菌で、酸素発生型の光合成を行うシアノバクテリアおよび酸素非発生型の光合成を行う紅色細菌、緑色細菌などがあります。紅色光合成細菌において、光エネルギーは光合成反応中心と呼ばれる膜タンパク質複合体に結合する光合成色素により電荷分離反応を起こし、光合成電子伝達反応が開始されます。
2)高電位鉄イオウタンパク質(HiPIP)
紅色光合成細菌において反応中心複合体への電子運搬を担う水溶性のタンパク質。タンパク質分子内部に、4個の鉄と4個のイオウからなる「鉄イオウクラスター」と呼ばれる金属補因子を結合しています。同じ鉄イオウクラスターを結合するタンパク質(フェレドキシン)に比べ、電子伝達反応に影響する酸化還元電位が高いことから「高電位」と名付けられています。
また、HiPIPは微生物の1種である光合成細菌において、太陽の光エネルギー変換に関わるタンパク質複合体である光合成反応中心に電子を渡す役割を担っています。光合成は生命の根源とも呼べる反応で、その重要性から光合成反応中心の立体構造決定は1988年にノーベル化学賞を授与されています。
3)結晶構造解析
分子の詳細な立体構造を決定するための実験手法。結晶試料に中性子やX線などのビームを照射し、結晶によって回折(散乱)されたビームを、検出器を用いて測定し、その回折強度分布から計算によって分子の立体構造を導くことができます。中性子とX線を用いた結晶構造解析をそれぞれ「中性子結晶構造解析」と「X線結晶構造解析」と言います。X線は原子の有する電子と相互作用するため、水素のような原子番号が小さく電子数が少ない原子からのX線回折強度は弱く、水素原子位置の観察が困難です。一方、中性子は原子核と相互作用し、回折強度は相互作用の強さを反映するため、タンパク質を構成する他の元素(炭素、窒素、酸素)と遜色なく水素原子を観察できます。
4)ペプチド結合
2つのa-アミノ酸の片方のアミノ基(-NH2)ともう一方のカルボキシル基(-COOH)が脱水縮合してできる結合です。タンパク質は20種類のアミノ酸がペプチド結合により連結した高分子で、基本単位であるペプチド結合はタンパク質が機能するために、立体構造を形成する際やタンパク質の安定化などに関与しています。
5)原子核散乱長密度分布
中性子結晶構造解析において、実験データの回折強度分布から計算される分子中の原子核の存在位置を示す密度分布です。原子核の位置で密度分布が高くなるため、散乱長密度分布に合うように分子モデルを構築していきます。
6)静電力
電気的に同じ性質を持った原子が近づくと反発しあい、異なる性質を持った原子が近づくと引き合います。この時に発生する電気的な力を静電力と呼びます。タンパク質分子中では、プラスの電荷をもつ塩基性アミノ酸とマイナスの電荷をもつ酸性アミノ酸との間で形成される塩橋や、水素結合なども静電力が関係しています。
7)鉄イオウクラスター
鉄原子とイオウ原子から構成される化合物で、2個の鉄と2個のイオウからなる2鉄2イオウクラスターや4個の鉄と4個のイオウからなる4鉄4イオウクラスターなど、さまざまな種類が存在しています。鉄イオウクラスターはタンパク質分子内部に補因子として結合することで、光合成、呼吸、酵素反応、遺伝子代謝など様々な生体反応に関与しています。
